深度学习模型辅助高分辨率医学病理图像自动检测
发布时间:2022-10-03 07:00
摘 要
人工智能领域的突破已经对全球社会产生了重大影响。在大健康医疗领域,尤其是在诊断学科例如放射学和病理学,将AI应用到患者数据的前景非常广阔。近年来,人工智能辅助数字病理图像诊断逐渐取代传统的显微镜观测。与自然界图像不同,病理图像在深度学习领域存在几个典型的问题:染色差异问题、空间相关问题、图像级别标注高成本问题。为了降低病理图像染色差异对于模型泛化性的影响,来自上海交通大学和壁仞科技研究院的团队提出两种解决方案,两篇相关论文于近期分别发表于9月在新加坡举行的医学图像顶会MICCAI 2022及医学图像顶刊IEEE Transaction on Medical Imaging (TMI),本文将对这两篇论文进行深入解读。
研究背景介绍
人工智能领域的突破已经对全球社会产生了重大影响。在大健康医疗领域,尤其是在诊断学科例如放射学和病理学,将AI应用到患者数据的前景非常广阔。近年来,由于医学数据的极速增长,人工智能辅助数字病理图像诊断逐渐取代传统的显微镜观测,并且有望在不远的将来成为病理学诊断的主要方式。WSI(Whole Slide Image)形态学特征复杂且图像尺寸庞大(约100,000×100,000像素),属于需要大量芯片算力的典型AI应用场景。数据的增长使得基于数据驱动的深度学习算法在计算机视觉领域迅速发展,逐步取代性能有限且泛化性较差的传统图像处理算法。
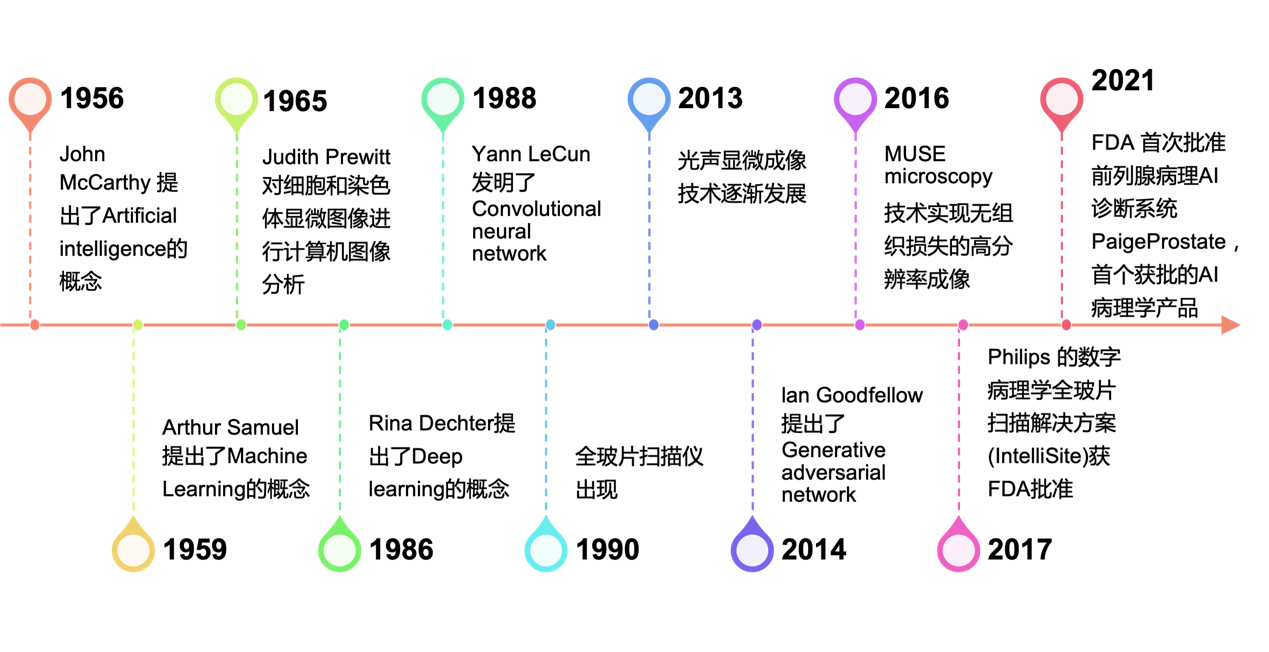
图1. 基于计算和人工智能图像分析的病理诊断里程碑[1]
图片来源:https://www.nature.com/articles/s41571-019-0252-y
本文解读病理图像的两篇论文,为上海交通大学与壁仞智能科技有限公司智能芯片与生态联合实验室一年多时间的共同研究成果。论文针对该类图像的特点,提出优化的神经网络算法。
与自然界图像不同,病理图像在深度学习领域存在以下几个典型的问题:
1.染色差异问题。WSI制片过程中,病理组织需要通过试剂染色来展现潜在结构模式,但不同医院染剂、扫描仪会使得电子化后的病理图像出现明显的染色差异,如图2所示。虽然有经验的人类医生专家并不会产生判断准确率下降的问题,但是由于训练和测试阶段数据分布差异过大,将会极大降低深度学习模型的泛化性。
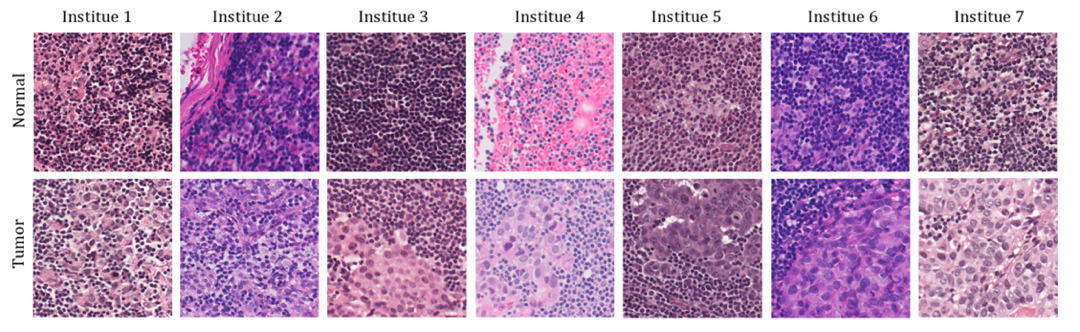
图2. CAMELYON16 数据集中来自多家医学中心的正常组织与癌症组织,不同组织之间染色风格各异[2]
图片来源:https://camelyon16.grand-challenge.org
2. 空间相关问题。受限于GPU显存,高分辨率WSI(如80,000×80,000像素)需要分割成小图像块patch(如512×512像素)才能作为深度学习模型的训练输入。但是在这种情况下,图像块层面的独立预测造成宏观层面的视觉上下文信息的丢失,使得模型在预测上缺失空间相关性,导致推理准确性降低,形成与先验知识违背的假阳性/假阴性。
3. 图像级别标注高成本问题。数据驱动的深度学习的方法需要大量详细准确标注的数据,以训练获得泛化能力强的模型。然而,病理图像由于自身的复杂性和专业性,特别是庞大的尺寸,图像块级别的精细标注需要消耗专家大量时间。
为了降低病理图像染色差异对于模型泛化性的影响,来自上海交通大学和壁仞科技研究院的团队提出两种解决方案。一是使用随机染色归一化实现染色增强的研究范式(Random Stain Normalization and Augmentation, RandStainNA)[3], 充分利用WSI空间相关性先验以提高模型推理性能,同时降低标注成本。二是提出病理图像可形变条件随机场方法(Pathology Deformable Conditional Random Field,PDCRF)[4],以空间自适应方式学习相邻上下文的偏移量和权重,以搜索高代表性图像块,用于专家标注及组织细胞分类任务。两篇相关论文于近期分别发表于9月在新加坡举行的医学图像顶会MICCAI 2022及医学图像顶刊IEEE Transaction on Medical Imaging (TMI)。
论文一
病理图像分析中选择染色归一化还是染色增强?RandStainNA实现两者统一。
Learning Stain-Agnostic Features from Histology Slides by Bridging Stain Augmentation and Normalization.
目前降低染色差异对深度学习的泛化误差主要有两个解决方案:染色归一化(stain normalization,SN)和染色增强(stain augmentation,SA)。染色归一化通过对其源图像染色分布到目标模板图像以减少染色变化,多种实证研究认为染色归一化是下游应用的必要前提。但是,染色归一化的精准定位能力很大程度上依赖于领域先验知识以进行模板图像的选择;另一方面,在一些情况下,比如联邦学习由于隐私正则化,中心处理器无法访问源图像,也就无法进行模板选择。
该论文发现了染色归一化与染色增强数学表示的一致性。染色归一化的转移来自先验Dirichlet分布,而染色增强通过一致分布变形图像。基于此发现,提出了RandStainNA,首次尝试将染色归一化和染色增强统一起来进行组织学图像分析。
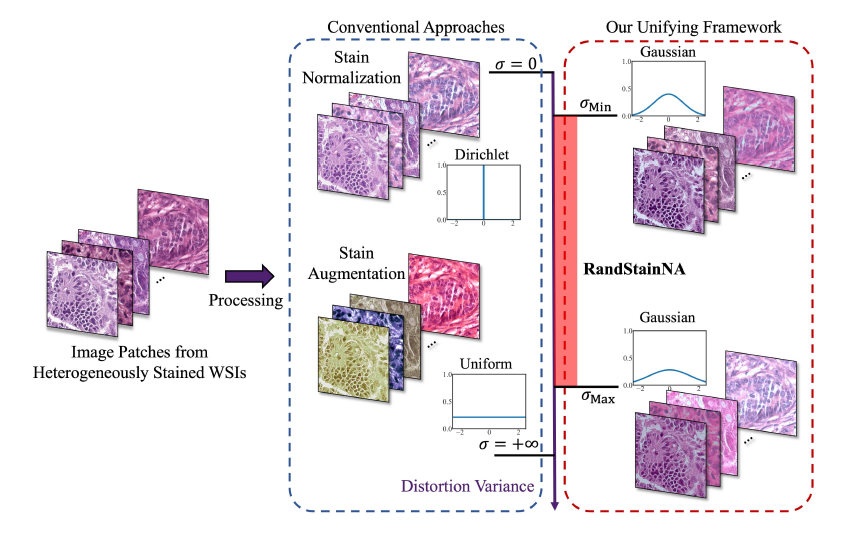
图3. RandStainNA整体方法示意图[3]
图片来源:https://link.springer.com/chapter/10.1007/978-3-031-16434-7_21
RandStainNA融合SN和SA以产生更具真实性的染色变种,根据引入随机颜色空间选择方案,将目标范围扩展到HED、HSV、LAB等多种颜色空间。

图4. RandStainNA融合SN和SA的特征并实现数学公式统一[3]
图片来源:https://link.springer.com/chapter/10.1007/978-3-031-16434-7_21
该框架通过预估计的染色风格分布自动分类出随机虚拟染色模板,即使用图像在颜色空间站三个通道分别的平均值与标准差的六个参数,而后使用这些参数对应的高斯分布迭代地随机生成虚拟模板。在参数估计时,我们并不使用整个训练集的平均值和标准差,而是使用更高效的随机挑选少量样本来计算参数,在不同训练epoch中使用变化较大的虚拟模板。而在训练阶段,通过虚拟模板对数据集进行图像层面的归一化,再转换回原来的RGB域后,即可实现分类或分割的下游任务。从SN视角在归一化的过程中引入了更丰富的染色风格,从SA视角增强的分别更加集中于一个受限的变形域中,使得框架具有普遍性而不局限于特定任务。
我们使用 NCT-CRC-HE-100K-NONORM 数据集用于训练,CRC-VAL-HE-7K数据集用于测试,分别包含数量为100,000和7180的组织学切片样本。训练时随机选取80%的样本进行训练,20%测试,在6种不同的模型下分类测试结果的比较展示如表1。实验表明,RandStainNA可持续提升准确率,超过单一的SN或SA。图5为不同方法的处理结果的可视化展示,验证RandStainNA可结合SN和SA的优点。图6为参数的UMAP编码比较,反映了我们在训练网络时可以很好地保证增强染色风格的真实性。同时,消融实验也证实了不同分布中高斯分布的准确率最高。在不同的样本规模下,使用这些参数也能够保持一致的准确率,拥有较好的鲁棒性。
表1 分类准确率测试结果比较[3]
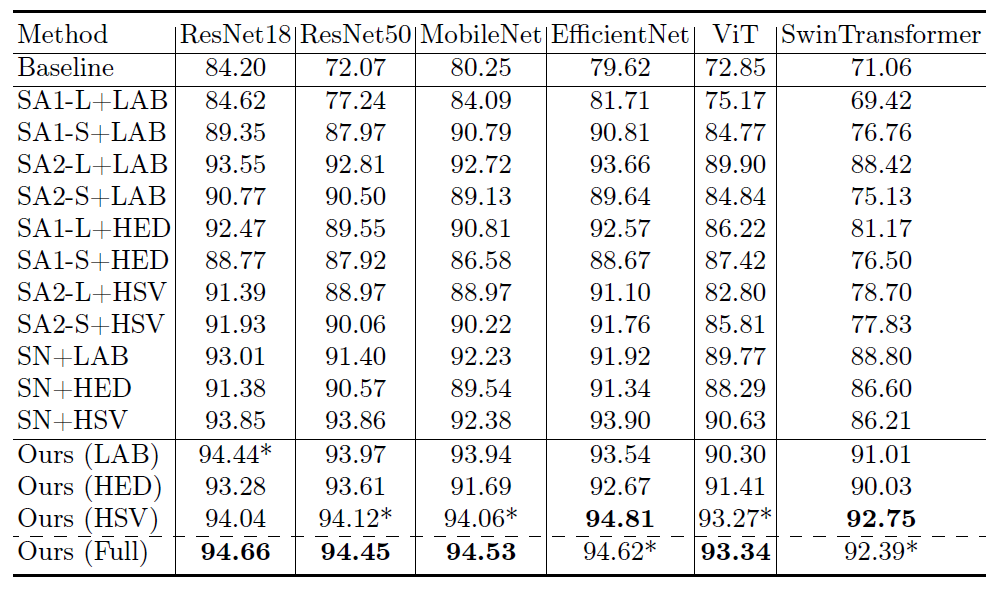
图片来源:https://link.springer.com/chapter/10.1007/978-3-031-16434-7_21
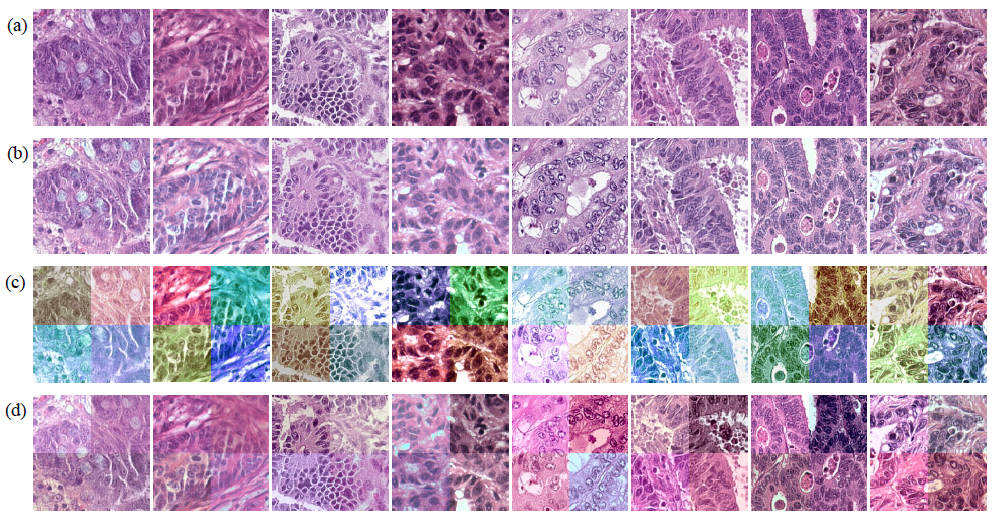
图5. 不同方法处理结果示例图:(a) 原始数据,(b) 染色归一化处理,(c) 染色增强处理,(d) RandStainNA处理结果图[3]
图片来源:https://link.springer.com/chapter/10.1007/978-3-031-16434-7_21

图6. 参数的UMAP编码比较可视化结果[3]
图片来源:https://link.springer.com/chapter/10.1007/978-3-031-16434-7_21
论文二
PDCRF将空间相关性的先验知识引入AI病理分析。
Identify Representative Samples by Conditional Random Field of Cancer Histology Images
使用基于图像patch的卷积神经网络(CNN)检测组织病理学图像中的异常组织时,上下文信息缺失严重。同样一个局部肿瘤区域,不同的神经网络输入patch的特征代表性差异非常显著。
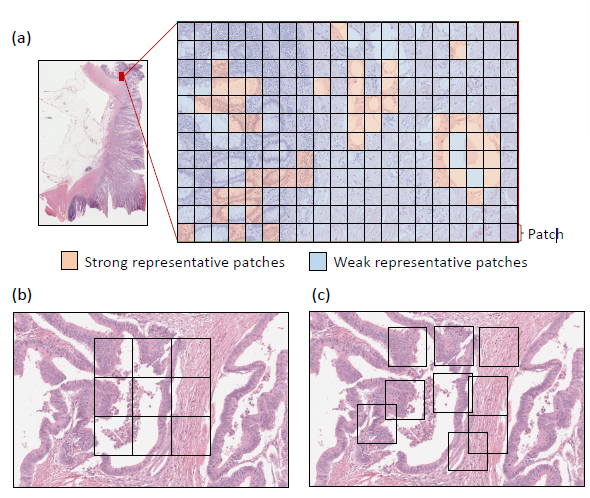
图7. 肿瘤区域中不同的神经网络输入图像具有不同程度的特征[4]
图片来源:https://ieeexplore.ieee.org/document/9855526/
基于相邻patch的不同代表性和空间关联性,本论文提出了一个通过评估组织学图像中的空间相关性来挖掘代表性图像块的框架,即病理图像条件随机场(PDCRF)模型。在执行常规分类任务之前,PDCRF根据其信息量动态定位感兴趣区域(ROI),然后识别和定位代表性图像块。此外,论文附加了一个主动学习框架,进一步证明模型有效性。该框架在训练集大幅度减少的条件下可维持同样的性能。
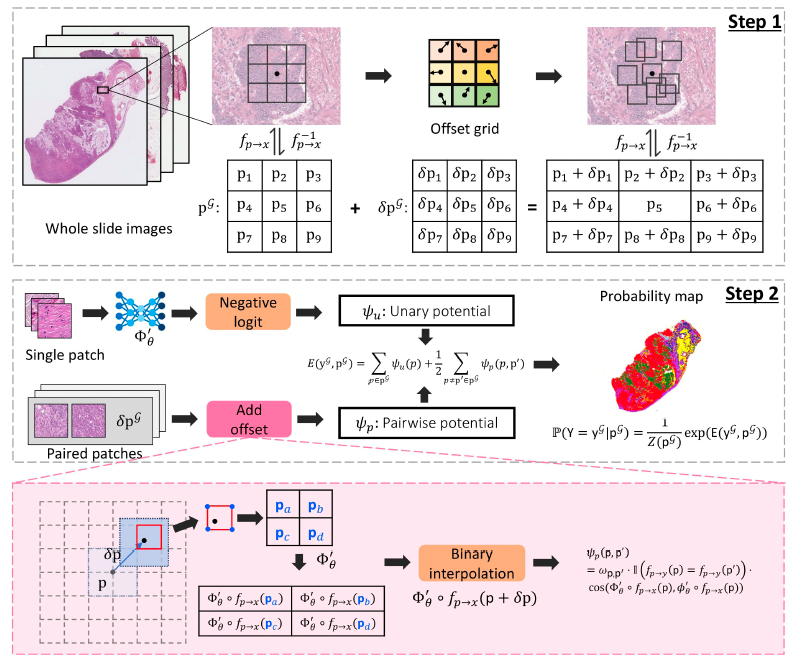
图8. PDCRF工作流程示意图[4]
图片来源:https://ieeexplore.ieee.org/document/9855526/
首先WSI采用常规分割策略裁剪出图像块patch,在ROI网格G中实现初始化。随后,逐步应用偏移量δp 的网格来迭代更新图像块集合 ,用于更多信息区域
,用于更多信息区域
PDCRF损失函数由三个独立部分构成,即分类模型 。PDCRF的输出是概率预测,随后将其分配给网格
。PDCRF的输出是概率预测,随后将其分配给网格 中的标签集合中的图像patch。对于一个给定的图像块级分类任务,我们应用标准原型交叉熵损失函数来训练这些参数,即:
中的标签集合中的图像patch。对于一个给定的图像块级分类任务,我们应用标准原型交叉熵损失函数来训练这些参数,即:

 是图像块
是图像块
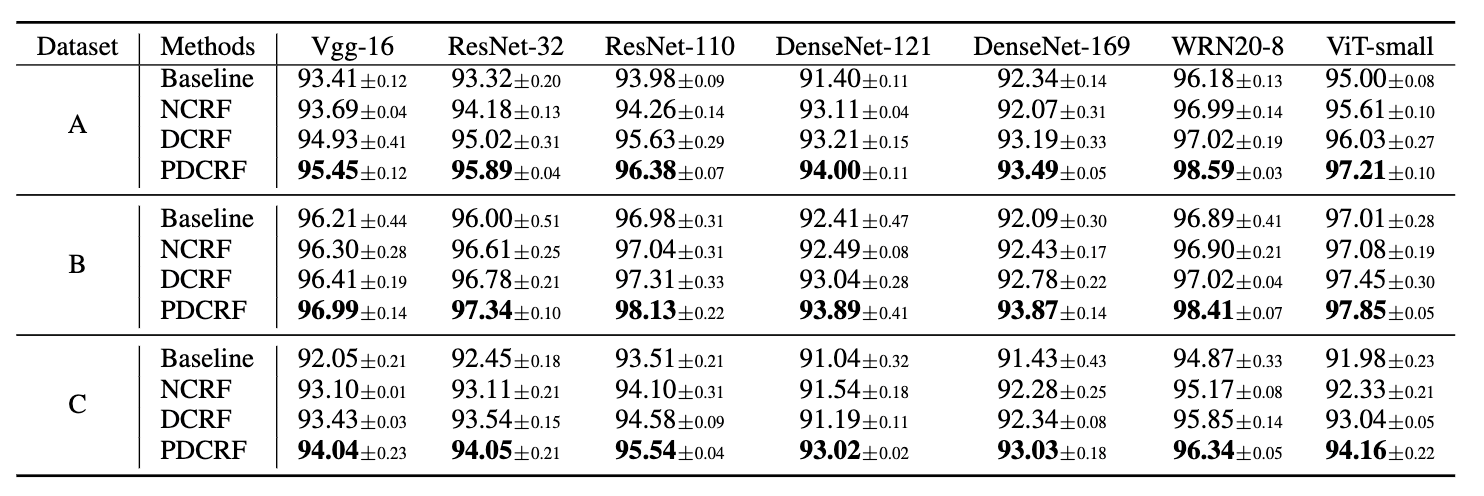
实验采用结直肠癌(CRC)组织的癌症组织样本数据来验证图像patch分类性能,包括三个TCGA公开数据集A:TCGA-STAD、B:TCGA-COAD和C:TCGA-READ。图像被分类为八类,用于图像块级分类任务。实验采用多种CNN和Transformer作为特征提取器,包括VGG-16、ResNet-32、ResNet-110、DenseNet-121、Denseet-169、WideResNet-20-8和ViT small。PDCRF与两种基于CRF的常见分类框架即Neural Conditional Random Field (NCRF)和Deformable Conditional Random Field (DCRF) 进行比较。准确度在数据集A、B和C上分别超出baseline 1.15-2.60%,0.78%-1.78%, 1.47-2.18%。
表2. NCRF, DCRF, PDCRF实验性能比较[4]
图片来源:https://ieeexplore.ieee.org/document/9855526/

图9. T-SNE图可视化展示不同方法的学习特征[4]:(a) 基准,(b) NCRF结果, (c) DCRF结果, (d) PDCRF结果展示
图片来源:https://ieeexplore.ieee.org/document/9855526/
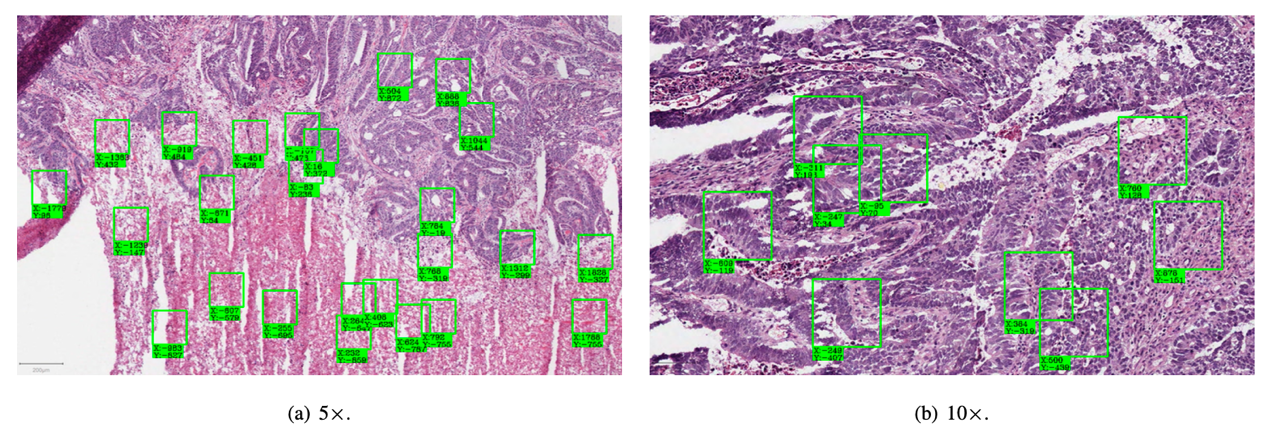
图10. 基于PDCRF定位强代表特征图像块patch,其中(a) (b) 分别为5倍和10倍图像放大[4]
图片来源:https://ieeexplore.ieee.org/document/9855526/
为了评估下游任务中的PDCRF,论文使用了包括三个主动学习方法,即1)随机抽样模型、2)熵抽样模型和3)纯损失预测模型。如图11所示,在多个不同尺度的训练集,PDCRF的准确率均在其他baseline之上。

图11. 主动学习任务中的性能比较[4]
图片来源:https://ieeexplore.ieee.org/document/9855526/
总 结
大健康领域特别是人工智能,被我国乃至全球发达国家都纳入未来发展的重点领域。医学数据的可用性及兼容性,新型计算架构的可解释性和鲁棒性,在过去几年已经取得了巨大的进展。但是整体而言,人工智能在健康领域的可靠性和实时性方面,依然等待提升,并处于落地的关键时期。随着算力的增强和算法的发展,临床推断的实时性将迅速提升,诊断的准确性也将逐步增强。
感兴趣的读者可以从我们的论文和代码中了解更多信息:
RandStainNA原文:https://arxiv.org/pdf/2206.12694
RandStainNA代码:https://github.com/yiqings/RandStainNA
PDCRF原文:https://ieeexplore.ieee.org/abstract/document/9855526
参考文献:
[1] Artificial intelligence in digital pathology—New tools for diagnosis and precision oncology, Nature Rev. Clin. Oncol., 2019.
上一个: 高性能Transformer推理分析
下一个: 让照片开口说话:AI驱动的人脸重演
近期文章





 商务合作:business@birentech.com
商务合作:business@birentech.com 业务电话:021-68773133
业务电话:021-68773133
